СО
Size: a a a
2020 June 24
При случае спрошу у коллеги, которая как раз H1B сейчас продлевает.
спасибо!
TF
спасибо!
Пока не за что, но вроде как 99%, что все ок, если уже находишься в стране. Иначе вся экономика и производство нехило так полетят
СО
Пока не за что, но вроде как 99%, что все ок, если уже находишься в стране. Иначе вся экономика и производство нехило так полетят
согласна, но кое-кого это уже на протяжении четырех лет кажется мало волнует
TF
согласна, но кое-кого это уже на протяжении четырех лет кажется мало волнует
Ну так уж совсем дури он пока не натворил, хотя и активно пытается. Ему просто не дают: как мне кажется, сложная, но надежная система сдержек и противовесов
СО
Ну так уж совсем дури он пока не натворил, хотя и активно пытается. Ему просто не дают: как мне кажется, сложная, но надежная система сдержек и противовесов
буду очень рада убедиться, что это так)
TF
буду очень рада убедиться, что это так)
Будем наблюдать)
AZ
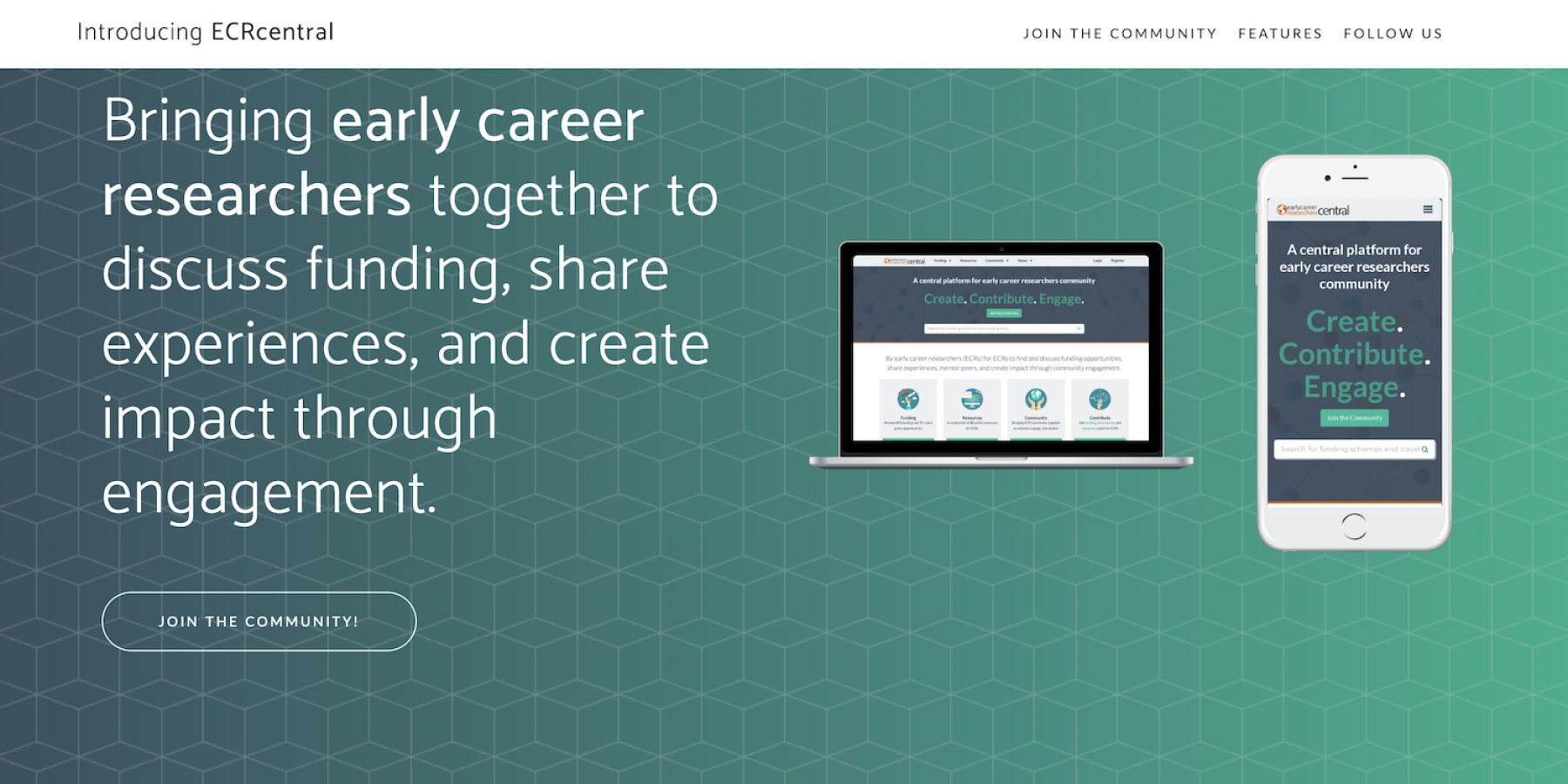
Кстати, тут еще один агрегатор грантов и стипендий подвезли: https://ecrcentral.org
DA
Возник возможно дурацкий вопрос к биоинформатикам.
Хочется отклассифицировать метагеномные короткие риды. Проблема в том что отклассифицировать надо не по стандартным базам а чисто по своим референсным последовательностям (среди которых есть и достаточно близкие). Кто это нормально умеет делать? Смотрим сейчас на бракен-кракен и centrifuge, а еще что разумное есть?
Хочется отклассифицировать метагеномные короткие риды. Проблема в том что отклассифицировать надо не по стандартным базам а чисто по своим референсным последовательностям (среди которых есть и достаточно близкие). Кто это нормально умеет делать? Смотрим сейчас на бракен-кракен и centrifuge, а еще что разумное есть?
AB
Возник возможно дурацкий вопрос к биоинформатикам.
Хочется отклассифицировать метагеномные короткие риды. Проблема в том что отклассифицировать надо не по стандартным базам а чисто по своим референсным последовательностям (среди которых есть и достаточно близкие). Кто это нормально умеет делать? Смотрим сейчас на бракен-кракен и centrifuge, а еще что разумное есть?
Хочется отклассифицировать метагеномные короткие риды. Проблема в том что отклассифицировать надо не по стандартным базам а чисто по своим референсным последовательностям (среди которых есть и достаточно близкие). Кто это нормально умеет делать? Смотрим сейчас на бракен-кракен и centrifuge, а еще что разумное есть?
А почему нужно еще что-то кроме кракена и центрифуги? В смысле чем они не устраивают?
DA
А почему нужно еще что-то кроме кракена и центрифуги? В смысле чем они не устраивают?
В кракене я не справился с нормальным созданием кастомных файлов с описанием таксономии (а без нее ничего не работает). Если это типа state of the art и самый простой способ - буду еще копаться конечно.
AB
таксономия вроде берется генбанковская, но сама я не делала
AB
надо @apredeus подождать, он делал
r
Возник возможно дурацкий вопрос к биоинформатикам.
Хочется отклассифицировать метагеномные короткие риды. Проблема в том что отклассифицировать надо не по стандартным базам а чисто по своим референсным последовательностям (среди которых есть и достаточно близкие). Кто это нормально умеет делать? Смотрим сейчас на бракен-кракен и centrifuge, а еще что разумное есть?
Хочется отклассифицировать метагеномные короткие риды. Проблема в том что отклассифицировать надо не по стандартным базам а чисто по своим референсным последовательностям (среди которых есть и достаточно близкие). Кто это нормально умеет делать? Смотрим сейчас на бракен-кракен и centrifuge, а еще что разумное есть?
Выравнивание ридов на свои референсы, потом разделить .sam на части по тому, на какие референсы они (риды) попали
r
В конце можно конвертировать разделенные .sam обратно в .fastq
DA
Выравнивание ридов на свои референсы, потом разделить .sam на части по тому, на какие референсы они (риды) попали
Я с этого начал) Там будут жуткие велосипеды если среди референсов есть похожие сиквенсы (а они есть в количестве)
r
Насколько похожие? Можно выравнивать в строгих условиях (без мисматчей)
DA
Насколько похожие? Можно выравнивать в строгих условиях (без мисматчей)
не поможет, куча неуникальных 99-меров даже
r
Как тогда (чисто физически) можно сепарировать риды, если они короче повторяющего в разных референсах 99-mer? Подозреваю, что никак
DA
(кстати, если это вопрос для какого-то более специализированного чата - буду рад перенаправлению)
O
Как тогда (чисто физически) можно сепарировать риды, если они короче повторяющего в разных референсах 99-mer? Подозреваю, что никак
Вроде обычно даже короткие риды длиннее 99?